Time for an update on last month’s progress!
LAB symposium
I attended the LAB symposium in Egmond aan Zee. Very interesting talks on a broad range of topics, from bacteriophages, to bacteriocins, to host-microbiota, to flavor and texture of lactic acid bacteria in food applications. I especially enjoyed the talk by Jens Walter who gave a broad overview of the natural history and lifestyle of the genus Lactobacillus. He mentioned that Lactobacillus iners, with the smallest genome amngst the lactobacilli, was “on its way to become an obligate symbiont”, given its loss of genes and functions. In the paper that was connected to this talk it says that the lifestyle-associated traits L. iners has “Fe-S—defense against peroxide, glycogen fermentation, adhesion”. However, I know of no published evidence that L. iners is capable of glycogen fermentation. Perhaps it is anecdotal, or just based on the genome.
Unfortunately, there were very few presenters (both posters and talks) who gave attention to vaginal LAB. I am convinced that the enormous effect of lactobacilli on reproductive health should in the future be a bigger part in the symposium and I am determined to create a lot of insight in the mechanisms underlying this colonization.
The symposium came to an abrupt and tragic end on Thursday after it became clear that one of the Irish attendees had been involved in a tragic accident after a midnight swim. Everyone was shocked about this tremendous loss in the LAB community. His name was Alan Lucid, may his memory be a blessing to his family and loved ones.
Growth of vaginal lactobacilli on glycogen
I have been growing L. iners myself, and have some preliminary indication that L. iners can grow well on glycogen, like Jens Walter wrote in his review. I also have a third replicate for L. crispatus growth on glycogen/water/glucose showing that this strain DSM 20584 can increase its cell density (grow?) on glycogen just as well as it can on glucose. (I updated the previous blog with the third replicate).
Organic acid analysis HPLC
Below you see the chromatogram of the UV detector of a L. crispatus supernatant after 24 hour of growth on NYCIII medium supplemented with either 0.5% glycogen, or water and L. iners grown on glycogen, and medium with glycogen as a control. I wanted to make sure that glycogen is indeed converted to lactic acid, and that these strains main product is lactate. At the moment I have only performed this analysis on one single experiment. This chromatogram indicates that lactate is the dominant organic acid produced by L. crispatus and L. iners. An acetate peak (expected at RT 18.9) is absent and also on the RID detector there are no big peaks detectable apart from some glucose in the medium and some lactate. I have not been able to identify all different peaks in the chromatograms. Especially the peaks at RT 21,8 and 23,9 have my interest since these might show some organic acid being produced and consumed, respectively.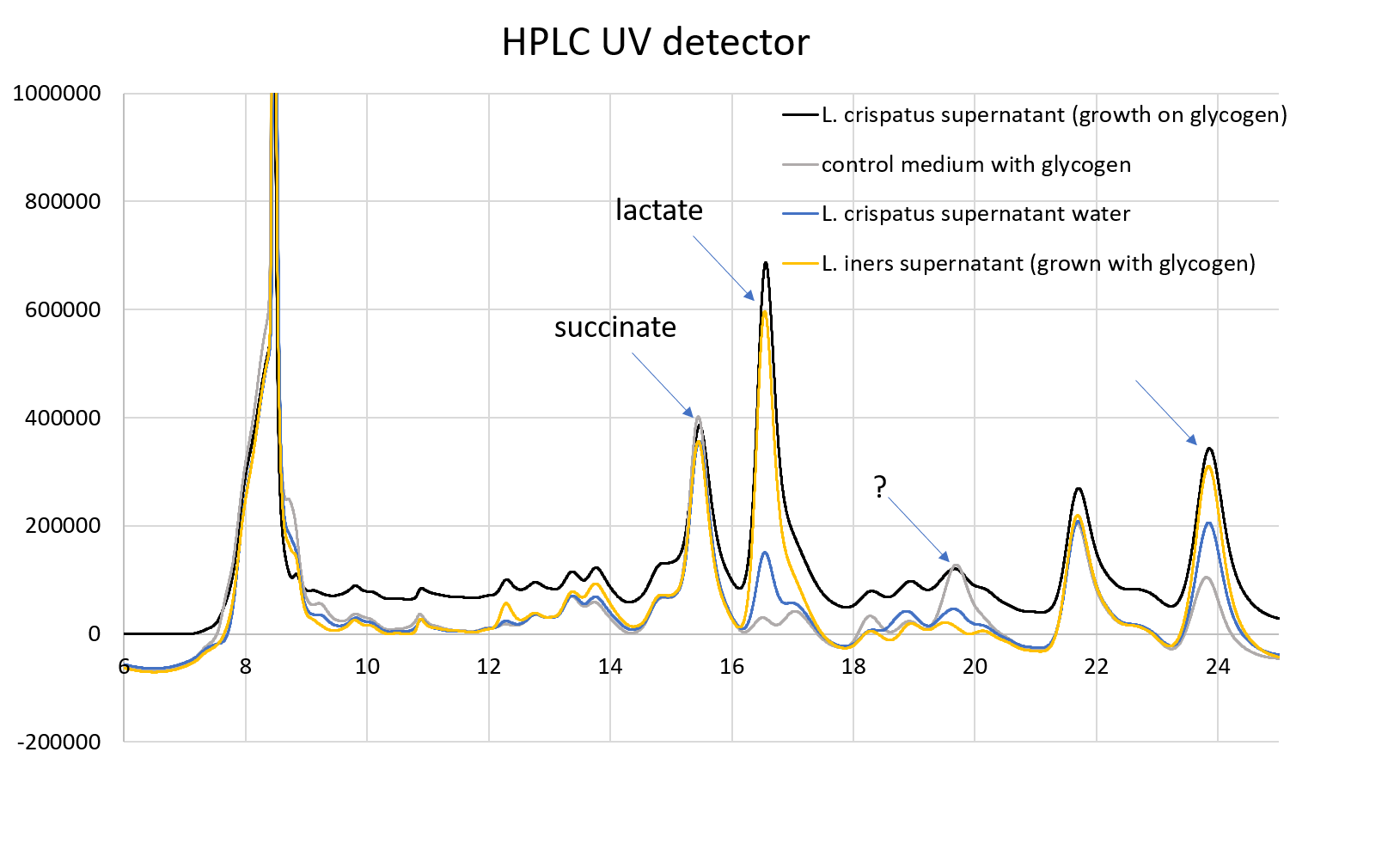
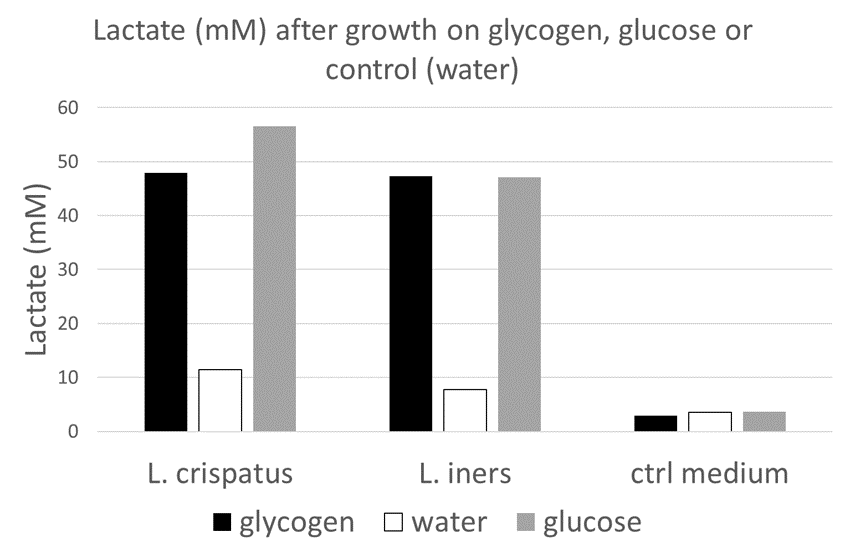
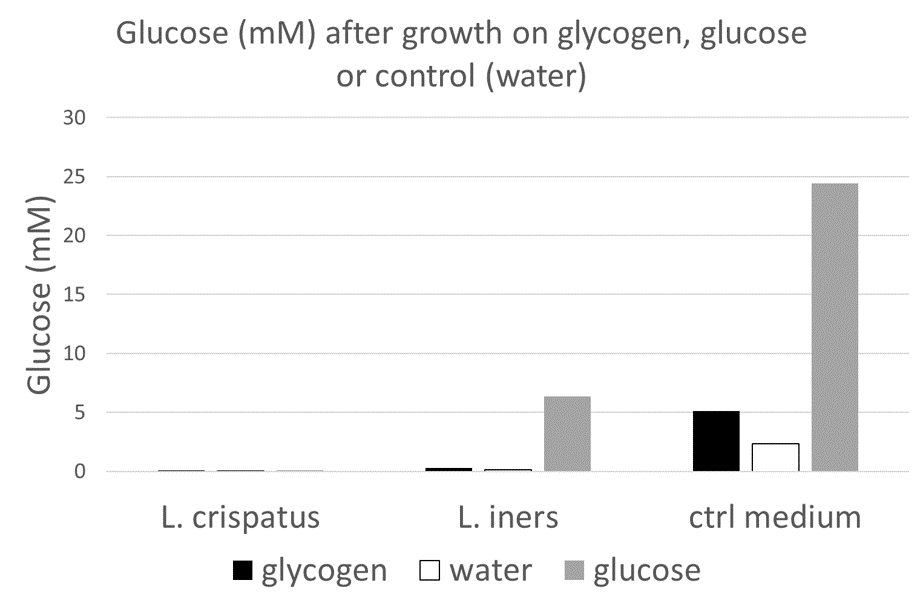
Below is an overview of lactate and glucose present in each supernatant. Beware, this is a single experiment, I will have to replicate these experiments!
Starch degradation
I continued using starch to detect any possible glycogen degradation activity in the supernatants. I believe I can conclude that supernatants of L. crispatus cultures that are grown on glycogen (and not on glucose or water) have starch degradation activity, but will only breakdown ~60* of a 1% starch solution.. Unfortunately, I do 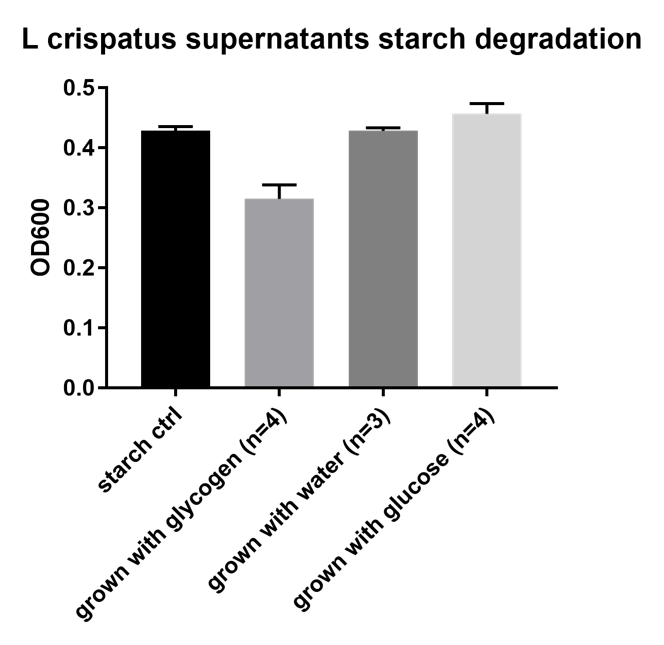
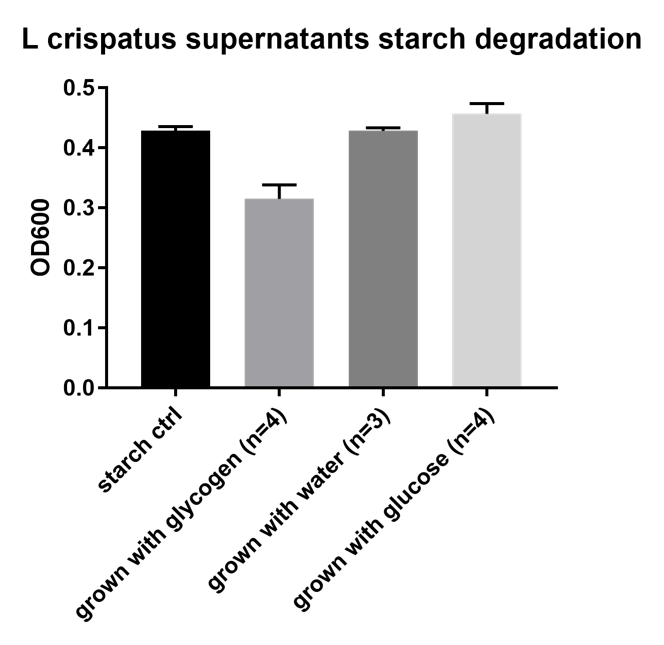 not have calibration curves for all measurements dates, so I am just showing optical density of the starch solution for now.
not have calibration curves for all measurements dates, so I am just showing optical density of the starch solution for now. 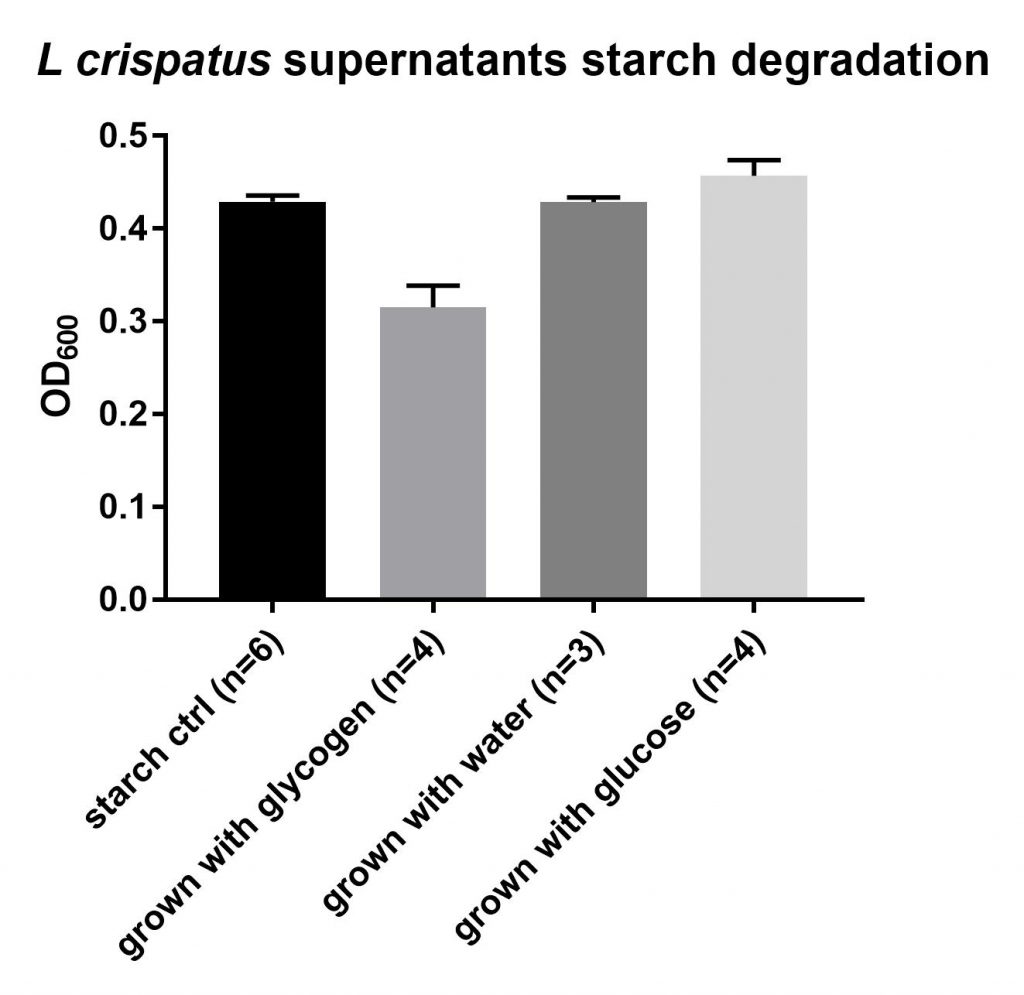
Preliminary results show that this activity is sensitive to a freeze/thaw cycle. It is also present in the pellet of L. crispatus and I have not been able to detect any degradation activity in L. iners supernatants (on glycogen/water/glucose). Lastly, in answer of the question posed in the previous blog (is amylase activity product inhibited): addition of glucose to the starch assay did not result in lower activity. These are only preliminary results and I am looking into these initial observations, I do not have sufficient replicates yet to be able to say anything with certainty.
Work discussion
On September 11th I gave a work discussion to the group here at the VU. Very excited to share the data from my postdoc in St Louis, and all new ideas. I also gave this presentation at a few different meetings of clinicians. I spoke at the regular morning gynecologists meeting at the academic hospital VUMC, where I was invited by dr. Nils Lambalk. I was also invited by dr Bing Thio and dr. Marinus van Praag to speak at the “Brugge Dagen”, the yearly meeting by Dermatologists of the ErasmusMC. Dermatologists in the Netherlands treat most vulvo-vaginal disorders. These talks for clinicians were in Dutch, I will upload an English version of this talk to Figshare at a later stage.